近年来,全基因组关联研究(GWAS)数据的公开共享推动了多族群因果推断研究,但地理环境与生活方式差异导致群体间遗传结构显著分化(群体分层),使GWAS汇总数据存在异质性。孟德尔随机化(MR)方法利用遗传变异作为工具变量(IV)推断暴露因素与结局的因果关系,其有效性依赖三个核心假设:关联性、可交换性与排他性。然而跨族群分析时,因遗传差异(如连锁不平衡结构变化),某一群体有效的IV可能在另一群体失效,加之社会环境因素对暴露-结局关系的修饰作用,导致因果效应在群体间不一致。
为探索跨群体恒定的"纯粹因果效应",传统策略分两步:先群体独立MR分析后元分析整合(方法假设限制多),或先融合GWAS数据再选IV(因LD差异可选IV有限)。两种方法均会累积统计误差,且元分析难以确保因果推断的群体普适性。本文提出单步整合方法MR-EILLS,基于环境不变性线性最小二乘法直接融合多源异质数据,实现:①兼容单/多变量场景;②允许IV违反可交换性/排他性假设;③输出无偏因果估计。相比传统MR与元分析,MR-EILLS显著提升估计精度、统计效能并降低错误率。
2025年8月18日,山东大学公共卫生学院医学数据学系侯蕾副研究员(本文独立第一作者)、生物统计学系陈昊助理研究员(本文通讯作者)和北京大学生物统计系周晓华教授(本文通讯作者)合作在Nature Communications上发表了题为“MR-EILLS: an invariance-based Mendelian randomization method integrating multiple heterogeneous GWAS summary datasets”的研究。本研究针对多源异质GWAS数据整合难题,创新提出基于环境不变性原理的孟德尔随机化方法MR-EILLS,构建跨种族普适的不变因果效应框架,同时支持单/多暴露分析,可处理违反可交换性与排他性的无效工具变量,确保理论无偏性与统计一致性,为异质遗传数据整合提供严格方法学基础。

本研究致力于解决多源异质性全基因组关联研究(GWAS)汇总数据整合所面临的核心挑战——遗传结构差异问题。来自不同祖先群体(如不同种族)的GWAS数据常因群体遗传背景、环境暴露、连锁不平衡模式等差异导致传统分析方法出现偏倚或效力损失。为此,作者创新性地提出了基于环境不变性原理(Environment Invariance)的孟德尔随机化方法,命名为MR-EILLS (Environment Invariant Linear Least Squares),旨在为跨种族的因果推断提供更为普适和稳健的解决方案。
MR-EILLS方法的核心创新在于构建了一个统一的统计建模框架,该框架基于环境(或背景)不变性的原理。其理论基石是,真正稳健的因果效应应能在不同的环境(这里特指不同的祖先背景)中保持相对恒定。该方法通过精密的模型设定和估计策略,系统性地识别并利用了这种跨越不同祖先群体的、对环境变化具有不变性的因果效应模式,从而建立起一个普适的不变因果效应估计框架。

图1 MR-EILLS框架
这一方法在研究实践中展现出显著优势,尤其在包含非洲、东亚、南亚、西班牙/拉丁裔、欧洲五大代表性种族的GWAS数据整合分析中取得了重要突破。其核心优势具体体现在三个方面:首先,方法学设计上同时支持单暴露(单一风险因素)及多暴露(多个相关风险因素)的复杂场景分析,大大扩展了应用范围。其次,能有效处理现实中普遍存在的无效工具变量问题,即使这些工具变量违反了传统孟德尔随机化严格依赖的可交换性(Exchangeability)假设或排他性约束(Exclusion Restriction),MR-EILLS也能通过其核心算法进行稳健估计,显著提升了结果的可靠性。第三,通过严谨的理论推导与证明,作者确保了MR-EILLS估计量具有渐近无偏性和统计一致性,为异质遗传数据的整合分析提供了严格的方法学保障和理论基础。
为了充分验证MR-EILLS的性能与实用价值,研究团队进行了广泛而深入的大规模模拟实验和实证分析。模拟实验系统地评估了在不同遗传异质性水平、无效工具变量比例以及效应异质性设置下,MR-EILLS与传统主流方法(如固定效应和随机效应逆方差加权法IVW)的表现差异,结果一致显示MR-EILLS在估计精度(更小的偏差)、统计功效(更高的检出率)方面均有显著提升,并能将I类错误(假阳性率)严格控制在理论水平。
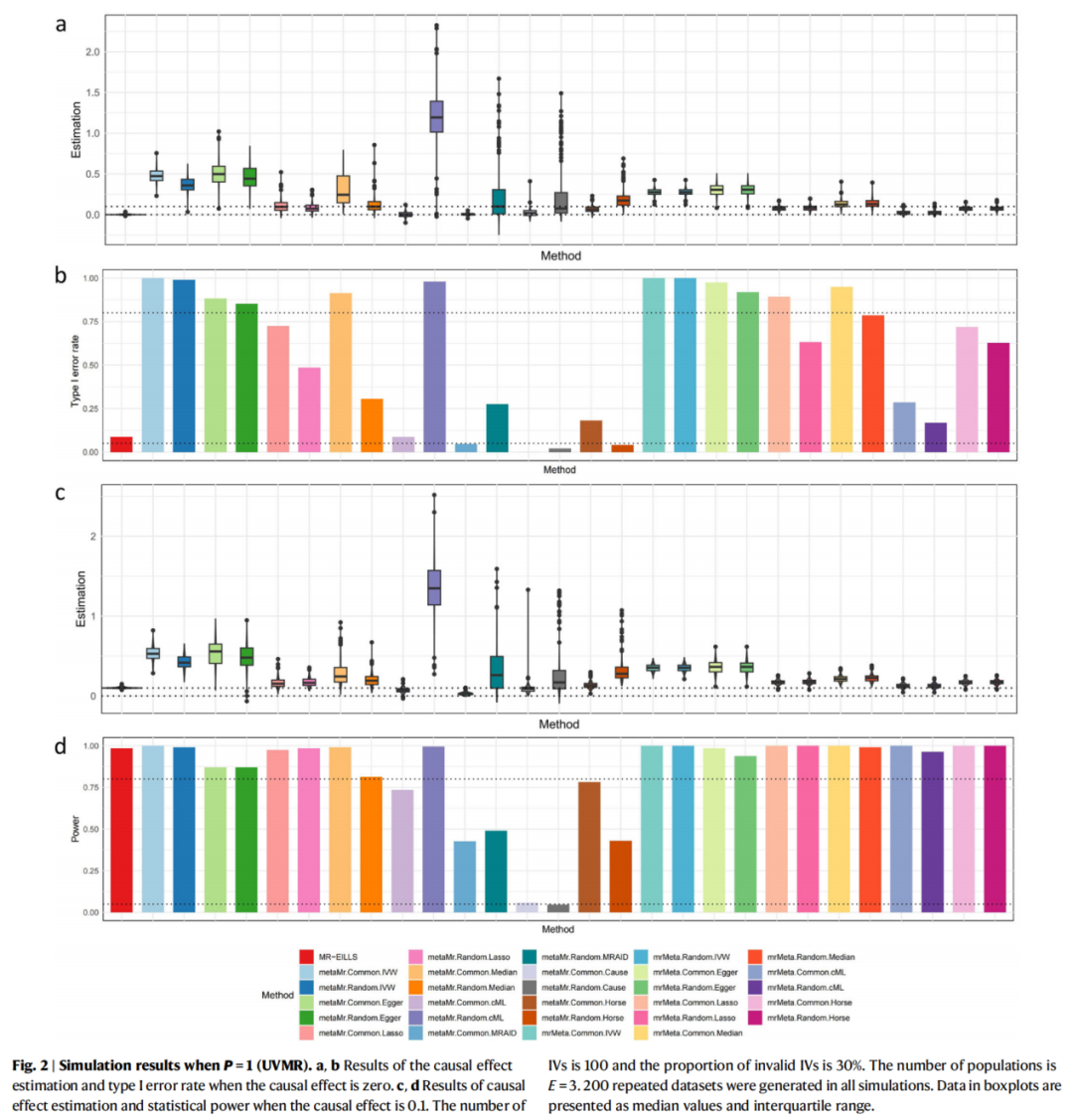
图2 部分模拟结果
在实证分析部分,作者将方法应用于11种血细胞指标与20种复杂疾病结局之间因果关系的系统探索。分析不仅成功验证了大部分既往研究已知的血细胞-疾病因果链(如血小板计数与心血管疾病风险),更重要的是,发现了多项在五大种族间显示出高度一致性的全新因果关系。这些跨种族验证的关联为理解相关表型的生物学机制提供了新视角,并为开发适用于多祖先人群的精准医学策略提供了更可靠的科学证据。
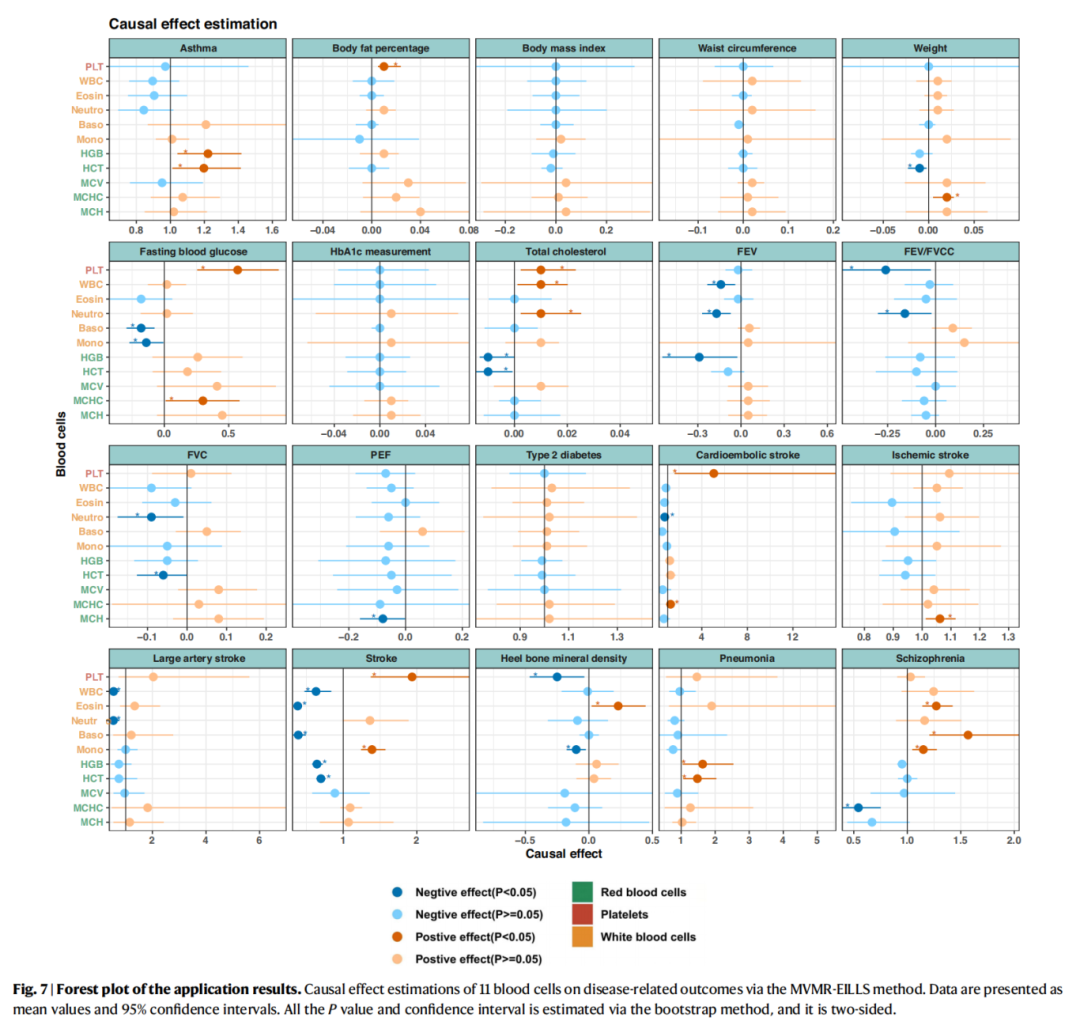
图3 实证分析结果
为促进方法学成果的快速转化应用并推动领域发展,研究团队已在GitHub平台(https://github.com/hhoulei/MREILLS)开源了MR-EILLS算法的完整实现代码,确保研究的可重复性。同时,同步发布了用户友好的R语言软件包,极大地方便了生物医学研究者和生物信息学家轻松集成该方法至其分析流程中,切实推动基于多祖先GWAS数据的复杂性状因果推断研究的进步。
图4 R软件包
文章结论与讨论,(未来)启发与展望
从更广泛的意义上看,MR-EILLS首次在数学本质上成功解耦了遗传异质性与无效工具变量造成的双重干扰问题。它通过建立对无效工具变量具有内在韧性的不变性估计框架,有效克服了群体结构差异带来的混杂影响。因此,MR-EILLS建立了一个适用于多元化、异质性族群GWAS数据集成的标准化因果推断新范式。这一方法学进步为多个研究方向开辟了新路径:在药物靶标验证方面,可为跨种族药物疗效和安全性的遗传证据提供更可靠支持;在跨组学整合分析领域(如整合GWAS、eQTL、pQTL等数据),为挖掘多层次的因果关系提供了更稳健的工具;在种族特异性生物学机制研究中,能更准确地区分普适效应与群体特异性效应。
通讯作者

周晓华
北京大学讲席教授,国家海外高层次人才计划特聘专家, 北京大学生物统计系系主任,北京国际数学研究中心生物统计及生物信息实验室主任,大数据分析与应用技术国家工程实验室数据处理及统计分析中心主任。国际生物统计学会中国分会理事长,中国现场统计研究会生物医学统计学会会长,中国数学会医学数学专委会主任委员,美国科学促进会会士,美国统计学会会士,数理统计研究院会士。曾荣获美国联邦政府退伍军人事务部研究生涯科学家奖、国际贝叶斯统计科学学会Mitchell奖,2022十大重庆科技创新年度人物。在顶尖统计学及生物统计学等期刊上发表SCI论文290余篇。曾主持多项国自然重点、国家重点研发计划,及国家发改委项目 . 出版了诊断医学方面的第一本综合性统计学著作Statistical Methods in Diagnostic Medicine(中文译名《诊断医学统计学》),由Wiley&Sons于2002年出版,第二版由同一出版商于2011年出版。

陈昊
统计学博士,助理研究员,入选山东大学青年学者未来计划、山东省青年科技人才托举工程,获北京大学博雅博士后称号(合作导师:周晓华教授),博士后出站后加入山东大学公共卫生学院生物统计系,主要研究方向为:高维数据统计分析、网络数据统计方法、多组学数据整合分析及因果推断方法。主持国家自然科学基金、博士后面上基金等,参与国家重点研发计划项目(骨干成员)、国家自然科学基金重点项目两项。近五年,以第一/通讯身份在国际顶尖的统计学及生物统计学Annals of Applied Statistics、Biostatistics、Statistics in Medicine等期刊公开发表论文10余篇,学术论文合作者包括北京大学、复旦大学、山东大学、华盛顿大学圣路易斯、埃默里大学等。
第一作者

侯蕾
医学博士,山东大学公共卫生学院医学数据学系,副研究员,主要研究方向为遗传统计中的因果推断方法研究及应用,包括工具变量、迁移学习、因果图结构学习、因果中介分析、选择偏倚、因果不变预测、多中心因果推断等等。共发表 SCI 文章 30 余篇,其中以第一作者/共同第一作者 11 篇 ,发表在Nature Communications、The American Journal of Human Genetics、Briefings in bioinformatics、PLoS Genetics等高水平遗传统计学、数学与计算生物学以及医学应用期刊上,同时为 eBiomedicine、Journal of the American Heart Association 等期刊的 审稿人。曾获得中国博士后科学基金特别资助、面上资助、国资计划B档资助、北京大学博雅博士后资助(合作导师:周晓华教授),参与一项国家自然科学基金面上项目、一项北京市自然科学基金青年 项目,发明专利一项,一项科技成果被评为国际先进水平。
此论文受以下基金资助:Novo Nordisk A/S、国家重点研发计划(项目编号:2021YFF0901400)、国家自然科学基金青年项目(批准号:82404378)、国家自然科学基金专项项目(批准号:T2341018)、山东大学青年学者未来计划,以及山东省青年科技人才托举工程、山东省医学会临床科研基金、山东省重点研发计划(项目编号:2024CXPT085)
点击链接查看论文原文MR-EILLS: an invariance-based Mendelian randomization method integrating multiple heterogeneous GWAS summary datasets | Nature Communications
转载本网文章请注明出处